Base de Dados
A base de dados utilizada foi pima.csv, contendo 768 instâncias de mulheres indígenas da etnia norteamericana Pima testadas por diabetes.
A base de dados contém 8 atributos e 1 classe binária.
A base também contém diversas entradas nulas, representadas pelo valor numérico 0, que NÃO foram inputadas no treinamento do modelo, propositalmente. Isto garantiu a robustez do modelo, uma vez que a inputação arbitrária de dados poderia comprometer seu uso em um cenário real. Ademais, a inputação de dados não gerou uma melhora perceptível acima de 5% nas métricas de F1 Score e Perda. Portanto o tratamento de nulos foi: manter as entradas como estão.
As entradas da primeira coluna, referentes ao número de gestações tidas pela paciente
O processo de "flip" (inversão binária) da classe de saída foi testada, e não gerou quaisquer resultados significativos. Portanto, a base foi mantida como original, tendo as saídas {0: Não tem diabetes, 1: Tem diabetes}.
Modelo MLP
As ênfases de desenvolvimento da rede MLP foram:
Minimização de Falsos Negativos.
Alta capacidade de generalização.
Baixo valor de perda.
Código
O modelo MLP foi treinado seguindo o código demonstrado na sessão de Código.
Foram utilizadas as bibliotecas: NumPy, Pandas, SciKitLearn e MatPlotLib
O código passou por múltiplas revisões e ajustes impossíveis de serem documentadas neste relatório. Entretanto, o histórico de commits pode ser conferido no repositório do GitHub.
Fase 1
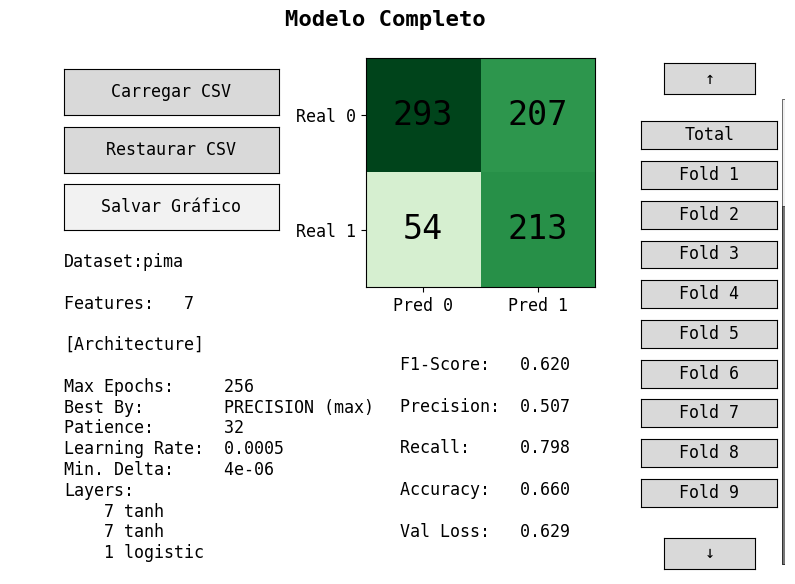
Figura 2: Melhor modelo em fase inicial obtido a partir de pima.csv
Na fase inicial de desenvolvimento, o modelo MLP foi treinado com o conjunto de dados pima.csv, focando em minimizar Falsos Negativos. Por razões médicas, esta métrica é a mais sensível, uma vez que classificar um paciente potencialmente diabético como não o sendo pode resultar em erros médicos graves.
Como podemos observar, a minimização agressiva de Falsos Negativos infere em uma redução da precisão do modelo como um todo. Isto prejudica a "correctividade" do modelo: o quão correto seu output é. Por causa disso, o modelo apresentado na Figura 2 foi descartado.
Fase 2
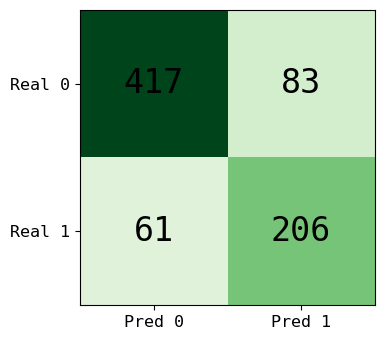
Figura 3: Melhor modelo obtido a partir do conjunto "stackado" (pima, pima-0n, pima-1n, pima-2n).csv
Figura 4: Matriz de Confusão gerada pela validação deste modelo contra a base de dados original (pima.csv)
Entretanto, a ferramenta permite que o usuário combine múltiplas instâncias da base de dados, como pode ser observado na Figura 3. Assim, a abordagem tomada na fase 2 foi de combinar repetidas entradas baseado na qualidade destas quanto à presença de nulos: quanto mais completa a entrada, maior a possibilidade desta estar repetida dentre as outras instâncias. A tolerância máxima permitida para repetição foi de 2 nulos por entrada.
Ainda assim, a base completa foi utilizada, gerando um modelo que "conhece" todas as possibilidades apresentadas na coleta de dados real. Mesmo com um Train/Test Split, a possibilidade de algumas folds treinadas passarem por um "overfitting" de entradas específicas era muito grande, e a capacidade de generalização foi posta em cheque, sendo pouco aferível.
Fase 3
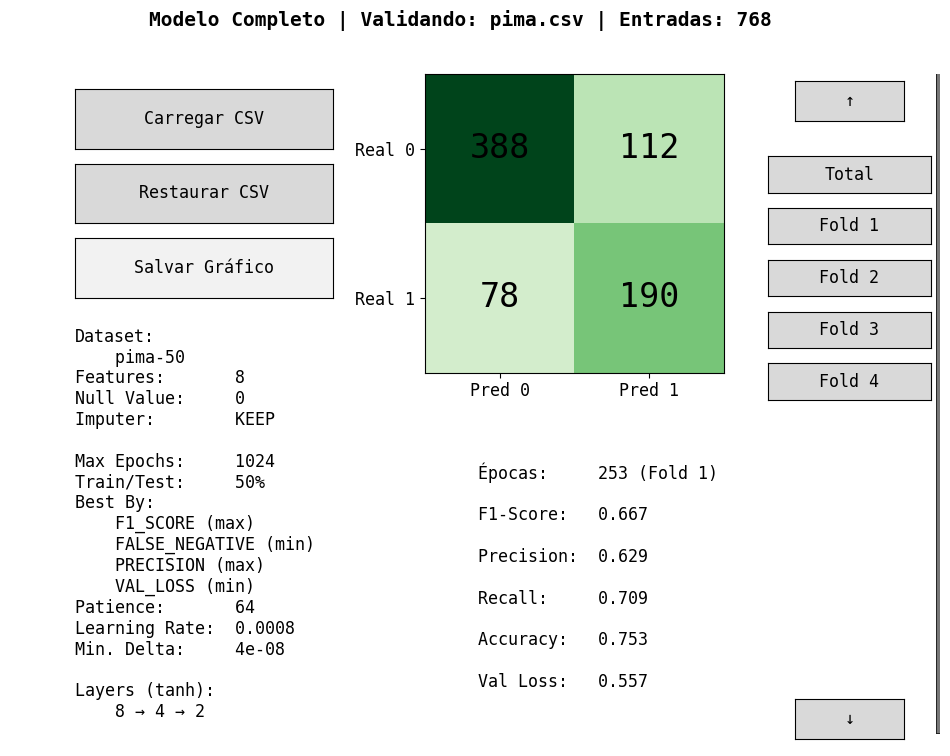
Figura 4: Melhor modelo em fase 2 obtido a partir de pima-50.csv
Figura 5: O mesmo modelo validado contra a base de dados completa e original (pima.csv).
Em uma terceira e última fase, o modelo foi treinado com uma base de dados reduzida em 50% do número de entradas (aleatoriamente, arbitrariamente). Esta base de dados foi salva como pima-50.csv.
Utilizando as métricas de arquitetura dispostas nas Figuras 4 e 5, podemos notar os seguintes pontos no modelo obtido através desta abordagem:
A base de dados utilizada durante Treino/Teste foi pima-50.csv, que contém somente 50% das instâncias originais de pima.csv. Ademais, um Train/Test split de 50% foi utilizado. Então realisticamente, o modelo só "conhece" 25% da base de dados.
Ao validar este modelo contra a base de dados completa (pima.csv), a proporção gerada na matriz de confusão bem como as métricas de F1 Score, Precision, Recall etc. mantém-se virtualmente idêntica (dentro de uma margem de erro calculada de ~3%). Isto indica uma ENORME capacidade de generalização.
Com isso, podemos concluir que o modelo obtido na fase 3 tem um grande potencial de generalização, minimizando falsos negativos dentro de qualquer conjunto de base de dados e contendo boas métricas de F1 Score e Perda.
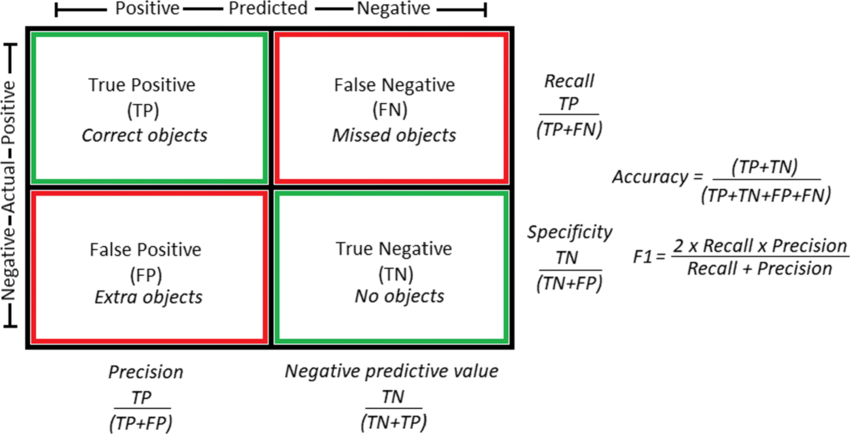 Figura 1: Matriz de Confusão de Modelos MLP (classificação binária) [Fonte: ResearchGate]
Figura 1: Matriz de Confusão de Modelos MLP (classificação binária) [Fonte: ResearchGate]